9−6:逆転写を介したファージtail fiberの変異
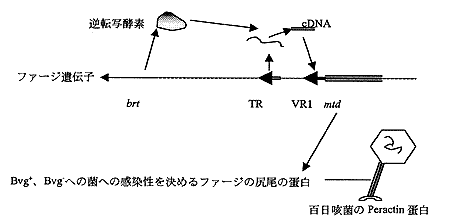
Bordetena pertussisは百日咳の起因菌である。気道に感染しそこで増殖しなければならない。しかし、その為には、菌は気道粘膜にしっかりへばり付く必要がある。つまり生体内で増殖するには、細菌の接着因子が重要な役割を果たす。一方、環境中での増殖にはそのような因子は不要である。そこで、百日咳菌はpertactinと云う接着因子を発現しているBvg+と云う相と、これを発現しないBvg-と云う相の間を行き来している。処で、pertactinはBPP-1と云うバクテリオファージのレセプターでもある。つまり、BPP-1ファージは病原性のあるBvg+相の菌には感染するが、病原性のないBvg-には感染しない。
BBP-1ファージをBvg-の菌に感染させると、少数ながら、感染が成立する変異ファージが出て来る。このような変異ファージにはBvg-の菌にしか感染しないものとBvg-、Bvg+両方の菌に感染するものとがある。ネズミチフス菌の相変異のようなf1ip-flopではない。
このようなファージでは何処に変異が起こっているのであろうか。調べると菌への吸着に使われる尻尾の蛋白Mtdにアミノ酸変異が起こっている。しかも、変異の場所は尻尾蛋白Mtd蛋白のC末44アミノ酸配列部分(遺伝子上では134bpのVR1領域)に限定され、変異するアミノ酸も22の特定な部位に限られている。非常に不思議な変異の起こり方である。
この変異には2つの遺伝子領域が関与していることが分かった。一つは、Mtd蛋白をコードする遺伝子(mtd)の下流にあるTR(temp1ate
repeat)と呼ばれる領域で、上記134bpのVR1領域と殆ど同じ塩基配列をしている。もう一つはbrtと云う領域で、原核細胞の逆転写酵素でレトロウイルスの逆転写酵素と相同な遺伝子である。brtを不活化するとファージの変異が起こらなくなる。恐らくbrt遺伝子産物がTRの転写産物を逆転写し、このcDNAが何らかのかたちでVR1領域に取り込まれ変異を起こすのであろう。
それでは、色々な変異はどうして起こるのであろうか?
変異の起こる22の場所はTRでは全てアデニン残基となっている。これらアデニンの部位で、逆転写の際に、誤った塩基を異常に取り込み易いか.或いは、塩基の修飾が起こる結果ではないかと思われる
(Science 295 2031-2032.2002)
9-7:生物界に於ける動き回る遺伝子
先に、IS、Tnのような細菌の染色体やプラスミド上を動き回る遺伝子について述べた。細菌、植物、ヒトを含む色々な動物のゲノム解析が進むにつれ、このような動き回る遺伝子は生物の遺伝子の中に氾濫していることが分かってきた。これは、C-value
paradoxと云うものと関係がある。
C-value
paradoxとは「生物の持つDNAの大きさが進化度と一致しない」と云う現象である。例えば、Amoeba proteusと云う原虫のDNAは29×1010塩基対もあるのに、ヒトは34×108であり、虫の100分の1の大きさしかない。ヒトの遺伝情報が原虫の100分の1であるとは考えにくい。恐らくは、原虫のDNAの大部分は有用な遺伝情報を持たないゴミ(junk)ではないかと云われてきた。
ゲノム解析により、このような推定が正しかったことが次第に明らかにされつつある。例えば、ヒトの
DNAの中で蛋白をコードしているのは全体の1.5%位で、遺伝子の数にして40,000位、少なくとも全体の45%は蛋白をコードしない動く遺伝子に由来するDNAである。植物のArabidopsis
thalianaになると、無駄な部分が少なくなり、DNAの39%が蛋白をコードし、遺伝子数は25,498とされている。他方、酵母では70%のDNAが蛋白をコードし(遺伝子数6340)、junk部分が少なく、無駄のない構造になっている。原核細胞になると更に無駄が無くなり85-95%がコード領域となる。多くの真核細胞生物は何故このように無駄なDNAを背負い込んでいるのだろうか。
9-7-1:真核細胞の動く遺伝子
動く遺伝子には、RNAへの転写とそれからのDNAへの逆転写を介するものと、細菌のISやTnのようにDNA分子として動き回る遺伝子がある。前者をレトロトランスポソン(retrotransposon)、後者をDNAトランスポソンと云う。
(1)レトロトランスポソン
レトロトランスポソンには、レトロウイルス同様LTRをゲノム両端に持つものとLTRを持たないものがある。前者をLTR transposon、後者をnon-LTR
transposonと云う。
LTRトランスポソンは、Ty1-copiaとTy3-gypsyの2群に分けられる。Ty1とTy3は酵母、copiaとgypsyはショウジョウバエ(Drosophila
melanogaster)で発見されたものである。図9-7-(1)に模式図を示す。Env遺伝子は、Ty1-copiaには無い。Ty3-gypsyにのみ存在する。

図9-7-(1)
Ty3-gypsyはEnvを持つので、レトロウイルス同様の伝搬形式を取ると思われるが、Ty1-copiaにも水平伝達のあることが証明されている。恐らく、Ty3-gypsyのEnvにより包み込まれウイルス粒子として別の種に感染すると思われる。Ty1-copiaタイプはEnv無しに一つの細胞の中で染色体上を動き回る事が出来る。しかし、Gag蛋白は持っている。この事はGag蛋白は動き回る為に必須であることを示しているのかも知れない。レトロウイルスのプロウイルス組み込み複合体がGag蛋白を含んでいる事を考えると、その可能性は高い。
LTRレトロトランスポソンの特徴は組み込み部位に特異性のある点である。酵母のTy1は、Pol
IIIで転写される遺伝子(tRNAと一部rRNA)の上流数百塩基の処に固まって組み込まれている。これは、酵母DNAの7割がコード領域であり、でたらめに組み込まれると酵母にとって致死的になりえる事
と関係があるかも知れない。実際Ty1の組み込み部位を調べてみると蛋白をコードする遺伝子の中に存在するものは1つもない。
図9-7-(2):Non-LTRレトロポソン
Non-LTRトランスポソンにはLINE (long interspersed nuclear
elements)とSINE (short interspersed nuclear elements)とがある。SINEにはヒトのAlu配列があり、ヒトゲノムの10.6%を占める(図9-7-(2))。
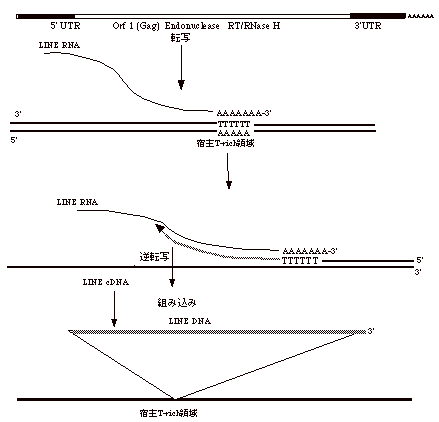
LINE は、RNAに転写されると組み込み複合体となり、そのEndonucleaseが染色体DNAのT-richな部分を切断し、3’
のpoly-A配列、(A)5-11、によりT-richな配列とhybridizeする。切断された染色体DNAのT-rich配列をprimerにしてそこからLINEゲノムを逆転写しcDNAを合成する。この特徴は、RNAからDNAへの逆転写がトランスポソンの組み込みとカップルしている点である。3’側から逆転写が起こるため、途中で転写が止まり、組み込まれたLINEゲノムは、5’端を欠損する場合が多い。
HTLV-1の組み込み部位を調べると半数近くが5’端を欠くプロウイルスである(Hiramatsu &
Yoshikura, J. Virol. 58, 508-512)。HTLV-1は典型的なLTRを持つレトロウイルスであるが、Non-LTRレトロポソンと共通した組み込み経路を持つのかも知れない。
(2)DNAトランスポソン
染色体 DNA上に存在するトランスポソン遺伝子が、RNAへの転写を介さずに、動き回る点で、細菌のISやTnに似ている。トランスポジションは、細菌のclass
Iトランスポソンと同じく切り出され、直接、宿主DNAに組み込まれるnon-replicative type
のものである。従って、トランスポソンDNAが切り出された後のDNAの修復が必要になり、これが無いとそこに二重鎖切断を残し、宿主にとって致命的になり得る。
Tc1/marinerトランスポソン
Tc1はC. elegansで、marinerはDrosophilaで見つかったトランスポソンである。DNAトランスポソンの中で最も単純な構造をしており、サイズも1.6-2.3kbと比較的小さい。ゲノムの両端に逆向き繰り返し配列(IR)があり、IRに挟まれてトラスポゼース遺伝子がある。従って、細菌のISによく似ている。Transpositionはトランスポソンが染色体から切り出され、新しい場所に組み込まれることにより起こる。組み込まれる部位の塩基配列は5’-TA-3’で、レトロウイルスの組み込みの場合と同様段違い切断(staggered
cut)が入る為、組み込まれたトランスポソンの両端に宿主染色体由来のAT配列が見られる。一方、トランスポソンが切り出された後の染色体部位は、blunt
end ligation(断端結合)や相同組み替えなどにより修復される。
P因子(P-element)はTc1/marinerの仲間である。この因子はhybrid
dysgenesisと云う現象を介して発見された。即ち、P因子を持つ雄のショウジョウバエとP因子を持たない雌を掛け合わせると子孫が出来ない。しかし、P因子を持つ雄をP因子を持つ雌に掛け合わせると子孫が出来る、と云う現象である。
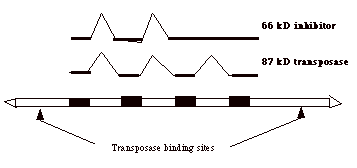
図9-7-(3):P因子とその転写産物
P因子の遺伝子の両端には31塩基対逆向き繰り返し構造があり、IRBPと云う宿主由来のトランスポジションに関与する蛋白が結合する。この蛋白は2重鎖DNA切断の修復に関与する酵素である。その内側にそれぞれtransposaseの結合部位がある。
P因子のTransposaseの遺伝子は、トランスポソンの真ん中にあり、3つのintron、即ち4つのexonからなる。
TransposaseのmRNAの完全なスプライシングは生殖細胞でのみ起こり、体細胞では不完全である。即ち、3番目のintronのsplice
outが起こらない。これは、体細胞ではこのスプライシングを押さえる因子PSIが発現している為である。この為、生殖細胞だけで活性のある87kDのtransposaseをコードするmRNAが転写され、生殖細胞だけでP因子遺伝子が飛び回れる。
一方、3番目のintronがsplice outされないmRNAは66kDの蛋白をコードし、恐らくtransposaseが結合する部位(両端IRの直ぐ内側にある)に競合的に結合するため、transposaseを抑制する。P因子を持つ雌の細胞ではこの66kD蛋白が多量に蓄積されている。そこで、P因子を持つ雌の卵に雄のDNAが導入されると、66kD蛋白が雄のP因子が発現するtransposaseを抑制する。一方、P因子を持たない雌との掛け合わせでは、卵には66kD蛋白は蓄積されていないので、P因子のtransposaseがそのまま活性化し盛んにtranspositionが起こる。結果、後の組み合わせでのみtranspositionによる染色体DNAの傷害が起こり、子孫が出来ないことになる。
hATファミリー
McClintockが発見したAc transposonがこの仲間に入る。11塩基対の逆向き繰り返し配列があり、その内側に4つのintronを持つtransposase遺伝子がある。遺伝子の大きさは4.6kbである。
Acの他に、縞模様のキンギョソウの花の原因であるTam、P因子同様hybrid dysgenesisを起こすhoboがある。切り出された後の染色体DNAがhairpinを作る特徴がある。免疫系の細胞でDNA再配列の際のRAG1、RAG2によるVDJ組み替えでhairpin構造が出来る点よく似ている。この為、獲得免疫に於けるDNA組み替えの起源は水平伝達により獲得されたtransposonにあるのではないかと云われている。
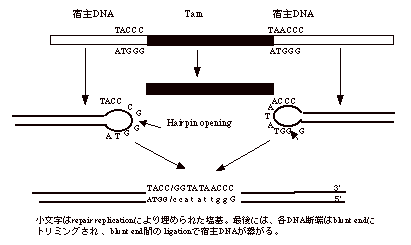
図9-7-(4):hTAファミリー
(3)トランスポソンの統合的な考え方
上に述べたように種々のトランスポジションがある。transposonの複製と組み込みが共役しているreplicativeなもの(Tn3のようなclass
II transposon)、ただ切り出され染色体の別の部位に挿入されるnon-replicativeなもの(P element、Ac
transposon等)がある。又、レトロウイルスの感染では組み込みの前駆体にはなり得ないと思われる環状プロウイルスが出来る。それぞれバラバラで何の共通性も無いように見える。
ここで注意したいのは、DNAが染色体 DNAに組み込まれる時、DNA transposonの場合にはtransposase、LTRレトロポソンの場合integrase、non-LTRレトロポソンの場合endonuclease等トランスポソンDNAとトランスポソンがコードする蛋白、宿主蛋白が複合体となったものが標的宿主DNAと相互作用をすると云うことである(図9-7-(5))。
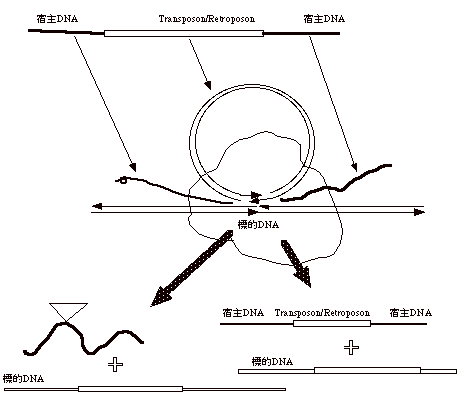
即ち、基本的にはtransposon
complexと云うべき複合体を介し、種々な組み替え産物が出来ている。この様な複合体の特徴は、組み換えに関与するDNA同士の境界部分、つまりお互いに組み換え合うDNA部分が、複合体の中で近接した状態になる事である。組み換え産物のかたちは、どのDNA鎖の断端と、どのDNA鎖の断端が連結されるかで決まる。その組み合わせを決めるのがtransposase或いはintegraseである。従って、基本的なメカニズムは種々のトランスポソン間で共通しているとも云える。
(4)
動き回るIntron
splicingには、真核細胞のmRNAの splicing(第6章参照)以外に、原核細胞やミトコンドリアで見られるGroup I、Group
IIのsplicingがある。前者、則ち、真核細胞のmRNAのsplicingは核内でRNAと蛋白の複合体spliceosomeにより行われin
vivoでのみ観察されるが、Group I splicing(テトラヒメナのrRNAで見られる)或いはGroup II
splicing(テトラヒメナのミトコンドリアmRNAで見られる)はin
vivoである事を必ずしも必要とせず、試験管内でRNA自身の酵素活性(Rybozyme活性)により起こる。
最近、このGroup I或いはGroup IIのintronの中にトランスポソンの様に動き回るintronのある事が分かってきた。このようなintronにはコード領域(open
reading frame, ORF)がある。ORFは、標的DNAに切り込みを入れるendonuclease、intron
RNAからDNAを逆転写するreverse transcriptase、RNA前駆体から特定のintron部分を切り出す酵素maturase、の遺伝子がある。
例えば、酵母にはω+とω—の二種類の株があり、前者にはgroup I intronがあるが後者にはない。交配すると子孫(haploidであることに注意)は必ずω+となる。則ち、交配によりω—株にgroup
I intronが移ったと考えられる。処が、ω+叉はω—の変異により、交配でω+とω—の子孫が同数出来るような変異株が見つかった。調べてみると、変異がω—の側にある場合の変異部位はintronが挿入される部位の傍であり、変異がω+の側にある場合には変異はintron内のコード領域(ORF)にあった。このORFはintronが挿入される標的DNAに2塩基の段違い切り込み(staggered
cut)を入れるendonucleaseをコードしていた。恐らく、intron
RNAを鋳型としてDNAが合成され、2つの切断部位の間に入り込むのであろう。挿入部位は一定なのでこれをintron homingと云う。
Group II intronはintronに特異性のある逆転写酵素をコードしていて、non-LTR
トランスポソンと同様な機構(6-7-1(1)参照)で組み込まれると考えられている。蛋白産物を含むリボ核酸蛋白と基質となるDNAを試験管内で反応させると、Group
II intronの基質DNAへの挿入が起こる。