上の実験で、注意して貰いたいのは、DNA合成が起こる場所は固定され、DNAの方が手繰り寄せられながら複製して行くと云う点である。DNA合成に関わる蛋白複合体が巨大な分子であることを考えると、話が良く分かる。転写の場合も同様で、RNAポリメラーゼがDNAの上を粘稠の細胞質のをかき分け動いて行くよりは、DNAを手繰り寄せてmRNAを合成していく方が合理的のように思われる。
DNAの複製点が隔壁の出来る場所に固定されているのはよいが、これだけでは複製した2つのDNAコピーが2つの娘細胞にそれぞれ分配されることを保障できない。真核細胞の染色体はcentromereに付いたkinetochorを介して細胞の両極に別れるが(図16-3-6-(5))、細菌にも似た機構のあることが分かった。
桿菌のBacillus subtilisでRacA変異(RecAではない!)は胞子形成の異常を来すが、その原因はDNAの配分異常である。RacA蛋白をChromatin
immunoprecipitationと云う技法で調べてみると、特にDNA複製中心に近い処にあるcentromere領域と結合した。Centromereに結合するRacAは、桿状をした菌の両極に局在するDivIVAに結合した。則ち、染色体のDNA複製開始点にあるcentromereはRacAを介し菌の両極にあるDivIVAに結合することになる。そうなると、2つに分裂した染色体はそれぞれ1ケ所で菌の両極に付いているので、細胞分裂が起これば、染色体も2つに別れることになる。
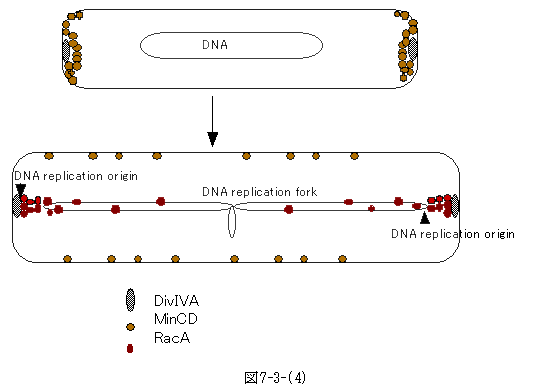
しかし、これでは、染色体の複製開始点は菌の極に付着したままになり、DNA複製に差し支える。これを制御しているのがMinCDで、菌が分裂に向けて成長している間は、MinCDがDivIVAの極在する部分に集まり、細胞分裂の開始を抑え、同時にRacAのDuvIVAへの結合も抑えるという仕組みとなっている。仮説の部分もあるが、Jacobの仮説への一つの回答である(S.Ben-Yuda,
DZ. Rudner , R. Losick, RacA, a bacterial protein that anchors chromosomes
to the cell poles, Science, 2003, 299, 532-536)。
7−4:細胞周期
細菌あるいは動物細胞の増殖は、DNAの複製と細胞分裂の2つの現象からなっている。
真核細胞では細胞分裂から細胞分裂までの事象、細胞周期、は、G1、S(DNA合成期)、G2、M(細胞分裂)が、この順序で正確に進む。M期が終わらずに次のS期に入る事はない。
細菌では、細胞分裂が済まないのに次のDNA合成が始まる。つまり、真核細胞では「細胞分裂をしていると云う」シグナルがDNA合成期へのコミットを抑え、DNA合成をしていると云うシグナルが細胞分裂を抑えているが、細菌では、そのようなシグナルによる細胞内の密接な調節はなく、増殖の速い菌では、細胞分裂とDNA合成が同時に起こる(図7-4-(1))。ただし、既に述べたように、DNA合成開始から一定時間後に細胞分裂が起こるので完全にDNA合成と細胞分裂が勝手に起こると云うことではない。
このような状況を反映し、真核細胞では数多くの遺伝子が細胞周期の調節に関与している。
図7-4-(2)は動物細胞の細胞周期を纏めた模式図である。細胞周期の調節には2つのチェックポイントがある。DNA合成に入るかどうか(つまりG1からS期への移行)と細胞分裂を始めるかどうか(G2からM期への移行)の2点である。