第6章 遺伝子とその発現
|
|
|
|
| 生物が生きていく為の全ての情報はDNA
にある。DNA は RNAに読み取られ、蛋白に翻訳される。同時に、その生物が生き残る為には、DNA
は正確に複製され子孫に伝えられなければならない。DNA
は遺伝子発現と複製の両面から考えなければならない。ここでは、前者を扱う。 細菌のRNA
合成、蛋白合成の概要を説明する。動物細胞(真核細胞)と基本的に違う点がある。
細菌には核がない。動物細胞には核がある。核は核膜で細胞質と分けられている。即ち細菌では核膜を挟んだDNA転写産物メッセンジャーRNA(mRNA)
の核細胞質移行の過程が無い。
この為、細菌では DNA合成、RNAへの転写、RNA
から蛋白への翻訳が同時に起こる(図6)。
DNA複製が盛んな菌では複製1ラウンド終わらない内に次のDNA合成が始まる。そのような状態では、DNA複製開始点(origin)に近い遺伝子はコピーが多くなる。この様な事を反映して、リボソームRNAや蛋白のように増殖の良い菌で大量の発現を要求される遺伝子はDNA複製開始点に近い部位にある。
動物細胞では、核内で転写された RNAは、改変を受けて細胞質へ送りだされる。核内の
RNAはDNA配列をそのまま反映しているが、細胞質に出るRNAは5'端に cap構造を持ち3'端にポリA配列を持つ。多くの遺伝子はintronを持ち、細胞質に輸送されたRNAはこれを失っている(splicing)。細胞質に輸送され蛋白合成の鋳型となる部分を
exon と云う。
一般的に云って動物細胞の mRNAはmonocistronicであるが、細菌のmRNAは
polycistronicである。即ち、動物細胞の mRNAは翻訳開始点が一つなので、1つの蛋白しかコード出来ない。
細菌細胞では、mRNAは複数の翻訳開始点を持ち、複数の蛋白をコードする。この複数の蛋白は、機能的に同時に発現される事が必要なものである。例えばラクトースの細胞への取り込みと分解を行う酵素群は1つのmRNAから翻訳される。転写調節により、必要な遺伝子群の発現を一度に調節出来る訳である。
このような転写単位をオペロン(operon)と云う。
動物細胞に感染するウイルスで、1本のmRNAしか出来ないウイルス、例えばポリオウイルスは、1種類の蛋白しか作れない。しかし、ウイルスは、ウイルス粒子蛋白、RNA複製酵素など複数の蛋白を必要とする。この場合、巨大な蛋白がまず出来て、それが必要な蛋白に切られていく(post-translation
cleavage)。
大腸菌では DNA 依存 RNA
ポリメラーゼは1種類である。真核細胞ではDNA 依存 RNA
ポリメラーゼは3種類ある。
PolI はrRNA(18S,5.8S,28S)を、PolII は mRNAを、PolIII は5SRNAとtRNA
を転写する。
|
|
 |
6-1:遺伝子発現の調節
細菌は環境に応じ、飢えるか、大宴会か、の生活を送っている。エサがあれば直ちに取り込んで利用し、無くなれば速やかに一切無駄なエネルギーを消耗しない窮乏生活に入らなければならない。
その代謝調節は大変効率よく且つ素早い筈である。一体どうやっているのだろうか?
代謝は酵素によって進行する訳だから、代謝の調節は言い替えると、酵素の合成あるいは活性の調節である。また、環境に速やかに対応すると云うことは、物質の取り込みとカップルした現象である筈である。
調節には種々の機構が知られている。まとめると次の様になる。
1.Stringent control
2.Genetic constitution
3.Control of operon expression
4.Regulation of enzyme activity
6-2:Stringent Control
細菌を新しい培地に植えると、一定の時間を置いて対数増殖期に入る。栄養や酸素を消費し尽くすと、増殖が止まり、静止期となる。栄養を加えれば、直ちに増殖が快復する。
この時、細胞のRNA量を調べると、細胞増殖とRNA量は平行して動いている事が分かる。静止期の培養に栄養を加えると、まず、RNA合成が回復し、次にDNA合成が始まる。このRNAの大部分はリボゾームRNAである(図6-2)。
DNA合成、RNA合成、蛋白合成におけるヌクレオチド或はアミノ酸が重合する速度は、細胞増殖の速度に拘らず一定である、従って総合成量は合成開始の頻度で決まる。
|
|
増殖の速い細胞はそれだけ沢山の蛋白を作らなければならない。その為には、沢山のリボゾームRNA(rRNA)
が出来、それが片っ端からmRNA
に乗っかり、片っ端から蛋白合成が起こればよい。(同時にRNA合成も片っ端から起これば更に蛋白合成も盛んになる。)エサがなくなったら蛋白合成などせず、じっと次のエサが来る迄待たなければならない。この為には直ちにrRNA合成を止める必要がある。このrRNA合成の調節が
stringent control である。
正常の菌に、この様な調節を司る遺伝子がある事は、この調節が無い変異菌(RelA-)が得られる事で分かる(図6-2)。 |
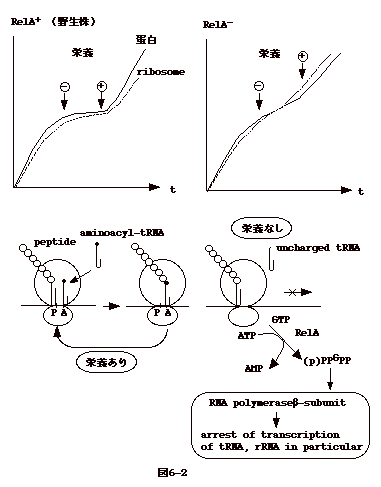 |
| stringent control を行う遺伝子は Rel A
と呼ばれる。エサが無くなると蛋白合成に必要なアミノ酸が欠乏する。そうすると、アミノ酸と結合しない
tRNA が増加し、蛋白合成中の ribosome のアミノ酸を受け取る A site
にアミノ酸の付いていない tRNA
がやって来る。これが引き金となって、(p)ppGpp が合成され RNA
ポリメラーゼの β-subunitに反応しこの活性をおさえる。その結果、まずtRNA,
rRNA の転写が止まる。RelA-
変異株では、エサが無くなっても (p)ppGpp が出来ずrRNA
合成が止まらない(図6-2)。
6-3:転写とDNA複製の速度
細胞増殖速度や転写産物の量に関わらず、次の値は、37度で一定である。
DNA: 800 nucleotides / sec
RNA: 80 nucleotides / sec
protein: 16 amino acids / ribosome / sec.
DNA複製の時、二重鎖DNAのsupercoilを解きながら動くgyrase の速度は100
supercoils/minである。相同組換えの際、RuvBが関与するbranch migration(後述)は10~20nucleotides/secである。
RNA合成の方がDNA合成よりのろい。そうするとDNA複製分岐点(replication
fork)がRNA polimeraseに追いつき得る。又、DNA合成方向とRNA合成方向が逆向きでぶつかる事もあるだろう。この様な時、どんな事が起こるのだろうか。
6-4:mRNAへ翻訳産物が結合する事による翻訳調節
ribosome はrRNA とリボゾーム蛋白 (r-protein)
から成る。いずれが沢山出来過ぎても効率が悪い。 |
|
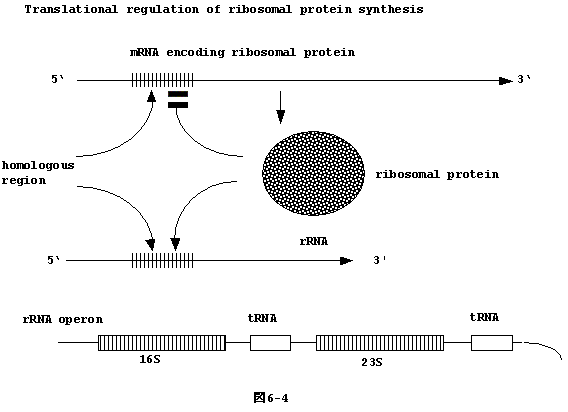 |
| rRNAの一部の塩基配列は r-protein
をコードしている mRNA の一部塩基配列と似ており (homologous)、r-protein
は rRNA, mRNA いずれとも結合出来る。従って、もし r-protein
が出来過ぎ、rRNAと結合出来なくなるとmRNAと結合しr-proteinの翻訳を抑える。これは、翻訳調節
(translational regulation)の良い例である(図6-4)。
6-5:遺伝子構造(Genetic Constitution)によるもの
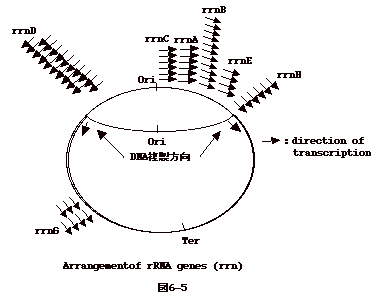
細菌では、DNA複製、RNA転写、蛋白への翻訳が同時に起こる。従って、DNA
合成を盛んにやっている菌では、DNA 複製開始点(Ori)
のすぐ下流の遺伝子は遥かDNA 複製終止点(Ter)
近くの遺伝子よりコピー数が多くなる。
大腸菌の遺伝子地図を見ると、一度に大量に合成される必要のある
rRNA, r-protein の遺伝子は、Ori
近くに集まっており、且、その転写方向はDNA
合成方向を向いている。つまり、複製点の進行と転写が衝突しないようになっている(図6-5)。 |
|
 |
6-6:Control of Operon Expression(オペロン発現調節)
この中には、次のものが含まれる。
1.Catabolite repression
2.Regulation by repressor or by inducer
3.Transcription regulation coupled with translation
4.Anti-termination
5.Choice of transcription initiation site σ factor
6.RNA stability
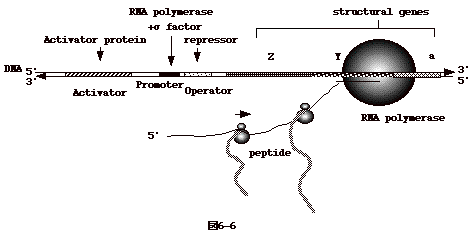
いずれも、蛋白をコードするmRNA
の発現量を調節する機構である。mRNA の転写は以下(図6-6)のように進行する。 |
|
 |
6-7:Lactose Operon
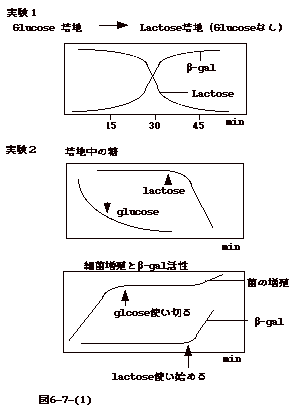
ラクトースオペロン(lactose operon)を例にしてcatabolite repressionとrepressor又はinducerによる調節を説明する(図6-7-(1))。
実験1.グルコース培地で飼っていた大腸菌をラクトースを含む培地に入れる。時間を追い、ラクトース量と
β-galactosidase ( lactoseをglucoseとgalactose
に加水分解する酵素)を量ると実験1のようになる。
実験2.上の実験で菌を植え込む培地をglucoseとlactose
両方を含むものに変える。同様に糖とβ-galactosidase (β-gal )の量を量る。実験2の様な結果になる。
実験1では、15分の lag の後β-gal が増加し始め60分で全菌蛋白の3%を占めるにいたる(これは、1,000倍の増加である)。β-galにより、lactose
はglucose と galactose に分解され、量が減って行く。つまり、lactose
が培地に加えらえた事でβ-gal が誘導 (induce)
される。従って、それまでは、β-galの発現が抑えられていた、と考えられる。実験2では、glucoseがある間は
lactose の利用が抑えられ、glucose が無くなって初めて lactoseが消費され始める。つまり、
glucose があると、lactoseがあってもβ-gal は induce されない。つまり
glucose はβ-gal の発現を抑える。glucose は lactose の代謝産物(catabolite)
なので、glucose存在下でβ-galの発現が抑えられていることをcatabolite
repression と云う。
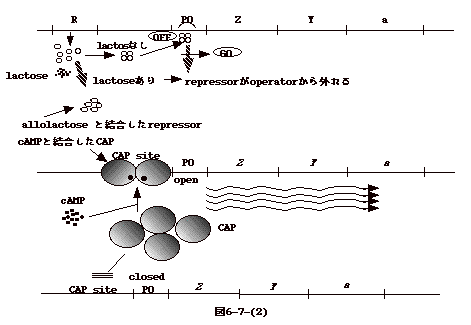
分子機構は以下の通りである(図6-7-(2))。
- lactose が無い状態では repressor は4量体となり operator に結合し RNA
polymerase のpromoter への接近を妨げる。結果、転写が押えられる。
- lactose を培地に入れると、lactose のisomer であるallolactoseとrepressor
が結合し、repressorはoperatorに結合出来なくなる。その結果、RNA
polymeraseはpromoter に結合し転写が可能となる。しかし、RNA polymeraseが転写を始めるには上流のCAP
siteにCAPという蛋白が付く必要がある。これが実験2に関係する。
即ち、
- glucose が存在すると、glucose
の取り込みにカップルして、リン酸化されたIIIg/c
が脱リン酸を受ける(図4-(6)参照)。
- ATP から cAMP を作るadenylate cyclase はリン酸化された IIIglc
を必要とする。glucose が取り込まれると、IIIglcは脱リン酸化されcAMP
が合成されない(図4-(7)参照)。
- lactose operon の promoter に RNA polymeraseがつくには、CAP (catabolite
activator protein ) とcAMPが結合しdimer になり、promoter 上流の CAP site
に結合する必要がある。
|
|
| 従って、glucose の無い場合のみ cAMP
濃度が上がり、CAPは cAMP と結合する。その結果 cAMPと結合したCAPはdimerを作り、これが
CAP site に付く。逆にglucoseがあるとcAMPが無くなり、CAPはCAP siteに付かず、lactose
operonはOFF になる。 この系は、物質の取り込みと、その物質の代謝に関係する遺伝子発現がカップルして調節されている良い例である。Catabolite
activator protein を介した転写調節を catabolite repressionと云う。 |
 |
 |
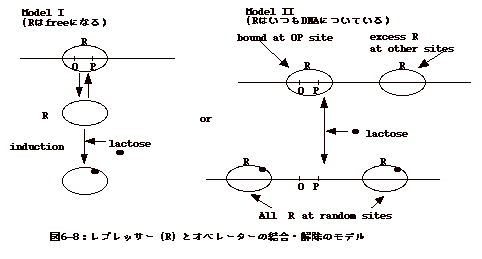
6-8:レプレッサーとオペレーターの結合・解離
|
| ラクトースオペロンの場合、lactoseを供給するとこれがレプレッサー(R)に結合し、lactoseの付いたレプレッサーはオペレーター(OP)に結合しなくなる。lactoseの供給が無くなるとlactoseはレプレッサーから外れ、レプレッサーはオペレーターに結合する。この機構によりlactoseが存在する時だけラクトースオペロンはONになる。これは、非常に効率のよい制御系である。 オペレーターに付いていない時レプレッサーはどこにいるのだろうか。もし、これが細胞質の中を自由にふらついているとすると、lactoseの供給が無くなり、lactoseがレプレッサーから外れても、直ぐオペレーターにくっ付
き、オペロンをオフにする訳には行かないのではないか、と云う疑問が起こる。そこで、lactoseの結合したレプレッサーは細胞質にフリーに漂っているのではなく、DNA鎖上にランダムにくっ付いているのではないか、と云う仮説が生まれた。
そこで、オペレーター部位とそれと同じサイズのそれ以外の部位へのレプレッサーの結合を調べた。レプレッサー単独の場合、オペレーターへの結合が2x1013(相対単位で、大きい値の方が結合が強い)であるのに対し、lactoseが付くとこの値が2x1010となる。一方、他のDNA部分への結合は、何れも2x106であった。従って、 レプレッサー単独の時のオペレーターへの結合特異性は
2x1013/2x106=107
であるのに対し、lactoseが付くと
2x1010/2x106 =104
となる。
lactoseが付いていても、レプレッサーはオペレーターにそれ以外の部分より104倍結合しやすい訳である。処が、或るDNA部分に付く確率は、該当部分のDNAの長さと結合のし易さの積に比例する。従って、オペレーター以外のDNA部分はオペレーター部位よりも104倍以上のサイズがあるので、lactoseが付いていてもレプレッサーが、オペレーター以外の部分より高い確率で結合することはない。即ち、lactoseが付くとレプレッサーの
オペレーターへの結合特異性は消失する。
このような実験からlactoseの付いたレプレッサーは常にDNA上をふらついているが、lactoseが外れるとオペレーターとの結合が高まり、結果として特異的にオペレーターに結合する事が推測されるに至った(B.
Levine, Genes IV354ー357頁)(図6-8)。 |
|
 |
6-9:アラビノースオペロン
アラビノースオペロンは、CAP蛋白によるCatabolite repressionを受け、且つ、アラビノース存在下でONになる、と云う点でラクトースオペロンと良く似ている。
しかし、次のような点で基本的に異なる性質のある事が分かった。
(1)araC遺伝子はアラビノースオペロン(AraABD)発現に必須であり、正の調節(inducer)をすると考えられた。処が
(2)アラビノースの存在に依存せずアラビノースオペロンをONにするようなAraC変異株を分離しAraCの部分diploidを作り、アラビノースによるオペロン誘導を調べると
野生型の表現型を示した。もしAraCがinducerであれば常にオペロンがONとなる変異AraCの表現型を示す筈である。
この様な、「理屈に合わない」点を解明しようとして研究がなされた。参考書として初めに紹介した本の著者、Schleifは以下に述べる現象の解明に大きな貢献をした人で、その研究の筋道が紹介した本に興味深く述べられている。
アラビノースオペロンの構造を図6-9の一番上に示してある。右に転写されるmRNAからラビノース分解に関わる酵素蛋白araB、A、Dが翻訳される。調節に関わるaraCは左側に転写される。
アラビノースが存在しない時には真ん中に示すような、DNAが折れ曲がった構造をとり、araC蛋白が2量体となって、araO2とI1に結合する。右側への転写は起こらない。 |
|
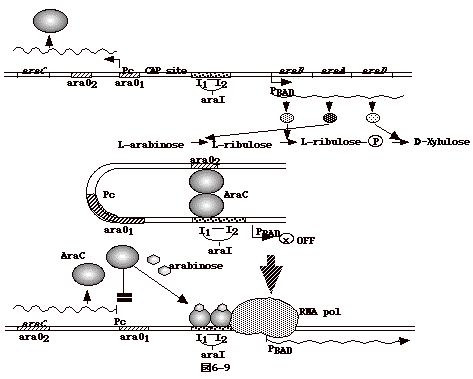 |
アラビノースを培養液に入れてやると、araCとアラビノースが結合し、araCの構造が変わり、I1とI2(araI)に図のように結合する。すると、RNAポリメラーゼが隣のプロモーター領域につきやすくなり、araBADmRNAが転写される。
araCは合成量が多くなると、自分自身のオペレーターに結合し、自分自身の転写をOFFにする。アラビノースが存在しない状態では、araCのプロモーター部分(Pc)はDNAの折れ曲がりの中にあり、恐らくその為、転写はOFFになっている。
CAP蛋白が結合する部分はaraO1とaraIの間にあり、グルコースの取り込みに連動したアデニレートシクラーゼ(adenylate
cyclase)の活性による制御を受ける。(グルコースがあるとcAMPがないのでCAPが結合せずアラビノースオペロンはOFFのままである。) アラビノースオペロンで特徴的なのは、DNAの折れ曲がりである。折れ曲がりの存在を示唆する実験的証拠として、I1とaraO2間に塩基対を挿入した実験がある。
DNAの1らせん回転分(helical turn)の10塩基対の挿入ではなんら影響を受けないのに、半らせん回転分に当たる5、或いは、15の塩基対を入れるとaraCによるrepression効果が無くなる事が観察されている(+5の法則と仮に呼ぶ)。半回転すると、折れ曲がった時、お互いに向き合う領域の一方が外側を向いてしまうのでrepressionの現象が無くなると解釈される。
真核細胞で転写効率を上げるエンハンサー蛋白がプロモーターから遥か離れたエンハンサー部位に付き、DNAの折れ曲がりによってエンハンサー蛋白がRNA合成に関わる蛋白をリクルートする場合にも+5の法則が存在するケースがある。
araCにアラビノースが結合すると、araCの構造変化が起こり、それによりDNAへの結合様式が変わる点は結果が逆なだけでラクトースオペロンのレプレッサーの場合と全く同じである。
又、アラビノース結合araC二量体がプロモーター上流のaraIに付きRNAポリメラーゼをプロモーター部位にリクルートする点は、後述の、ラムダファージにおけるレプレッサーcIが自分自身のオペレーター/プロモーター領域OR1、OR2に付きRNAポリメラーゼを付きやすくするのと良く似ている(図13-2-5参照)。
|
|
6-10:Regulation coupled with translation(翻訳連動転写調節)
|
| これは、細菌特有の現象と云ってよい。細菌では
mRNA への転写とそれからの翻訳は同じ場で同時進行する。 動物細胞では、核で
mRNA
が出来、それが細胞質に輸送されて翻訳が起こる。つまり、転写と翻訳の場が異なる。従って、翻訳が転写に影響を与える機会は無い。
細菌のアミノ酸合成に関わる operonであるtrp、 phe、 his、 thr、leu
等では、第一段階の調節として, lactose operonと同様 repressor
による調節がある。この場合は、それぞれ合成されるアミノ酸が
repressorと結合することによりrepressorがoperator
に結合できるようになり、転写が抑えられる。
しかし、これらの operonには第二段階の調節がある。これが翻訳に連動した転写調節である。
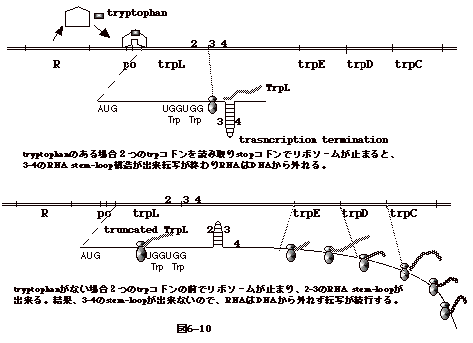
トリプトファン (trp) operon を例にとる。
promotor-operator に引き続き、トリプトファンをコードするtriplet
(UGG)が続く配列を持った短い読み取り枠 open reading frame (ORF)
があり、UGA のstop codonで終る。このORFをtrpLと呼ぶ。
トリプトファン合成に関与する酵素をコードする遺伝子trpE,trpD,
trpC, trpB, trpA は trpL の下流にある。もし、trpL
のすぐ下流で転写が止まると、トリプトファン合成酵素のコード領域は転写されないままになり、trpL
のみをコードする短いmRNA だけが出来ることになる。
tryptophan の存在下では、2つの trp コドンを読み取り stop
コドンでリボゾームが止まる。すると、3-4の RNA stem-loop
構造が出来、転写が終わり RNA は DNA から外れ、結果、trpL
のみをコードする mRNA が出来る(図6-10上)。
tryptophan がない場合、2つの trp
コドンの前でリボゾームが止まり、2-3のRNA stem-loop が出来る。
結果、3-4の stem-loop が出来ないので、 RNA は DNA
から外れず転写が続行する。
この時、 trpL の翻訳は trp
コドンの処で止まるが、原核細胞ではそれぞれの遺伝子(cistron)を独立に翻訳する内部翻訳開始点(internal
ribosomal entry site)があるので、下流の trpE , trpD
などは翻訳されることになる(図6-10下)。
トリプトファン合成酵素をコードする遺伝子 trpE、trpD、trpC
にはトリプトファン codon
は存在しない。従って、トリプトファンが培地中に無くても、トリプトファン合成酵素は出来る仕組みになっている。これは、転写の
anti-termination の一種である。以上はYanofskyが1979年に出した非常にエレガントな仮説である。
最近、B. subtilis
(枯草菌)のトリプトファンオペロンの解析が進み、トリプトファン存在下で転写が早期に停止し短いmRNAが外れる機構について、新しい事がわかった。即ち、tryptophan
attenuation protein (TRAP)の関与である。TRAPは75アミノ酸からなり、11量体を形成する。トリプトファンがこの11量体に結合すると、11量体TRAPはmRNAのトリプトファンコドンが11個並んだ部位に結合し、mRNAの2次構造を転写終結に至る構造に変える(Antson
et al, Nature, 401, 235-254, 1999)。 |
|
 |
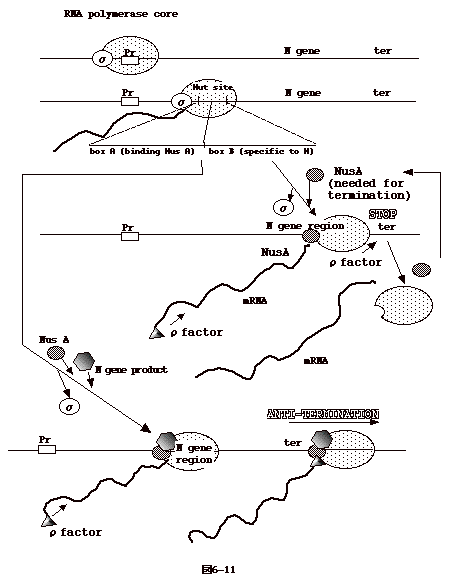
6-11:もう一つの転写終止阻害(antiterminatioin)
蛋 白とRNA polymeraseの相互作用を介したもう一つの anti-termination
がある。ラムダーファージでは、初期遺伝子の転写が済むとRNA
polymeraseは転写を止める。初期遺伝子であるN蛋白はこのRNA polymeraseと相互作用をして、RNA
polymeraseをさらに下流の転写を可能にする。
原核細胞での転写停止は次の2つの型がある。
ρ因子に依存しないもの
ρ因子に依存するもの
前者は転写が最点迄進むと mRNA の3'側に特徴的なヘアピン構造が出来てこれがシグナルとなりRNA
ポリメラーゼが RNA
から外れ、転写が止まるものである。トリプトファンオペロンのtrpL翻訳後の転写停止はこの例である。後者は転写停止にρ因子を必要とするものである。
N蛋白は、ρ因子依存性転写停止に関与する。RNAポリメラーゼが転写開始点を認識するにはσ因子が必要であるが、転写中にある部位に来るとσ因子は
宿主Nus蛋白と置き代る。転写終止に関わるρ因子は mRNA の5'からmRNA
に乗っかり3' 側の方に移動する。Nus蛋白- RNA
ポリメラーゼが転写終止点迄来ると、追いついて来たρ因子の作用により、転写複合体はDNAから外れる。
λファージのN蛋白はNus蛋白- RNA ポリメラーゼ複合体を nut部位で認識しN蛋白-
Nus蛋白- RNA
ポリメラーゼ複合体を作る。こうなると、ρ因子が来ても転写複合体は鋳型
DNAから外れず転写が続く。これが N蛋白による anti-termination
である(図6-11)。 |
|
 |
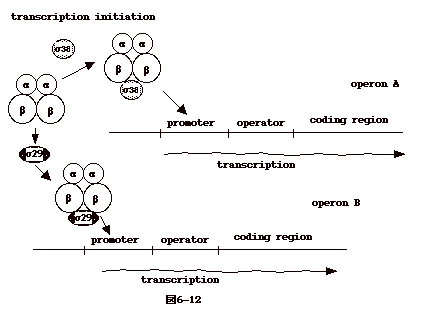
6-12:シグマ因子による転写operonの選択
シグマ因子( σfactor ) は RNA polymeraseとcomplex を作っており、どのpromoterを選ぶかを決める。細菌が胞子を作ったり、ファージ(細菌に感染するウイルス)が宿主遺伝子からファージ遺伝子の転写にスイッチを切り替える時など、大幅な代謝の変化をもたらす場合によく利用される。
Bacillus subtilisでは増殖時にはsigma 37を利用するが、胞子形成が始まると
sigma 29 にスイッチされる。また、この菌が SP01ファージに感染すると、ファージのコードする
sigma gp 28が RNA polymeraseを乗っ取る。このようにして、sigma
因子により読むべき遺伝子群が決められると、その遺伝子群しか読めなくなる。 |
|
 |
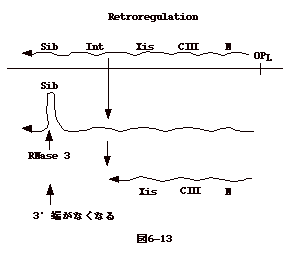
6-13:Retroregulation:mRNAの部分分解による発現調節
|
| mRNA
の安定性は遺伝子発現に大きな影響を与える。ラムダファージが宿主に溶菌的に感染すると(後述)、
N 遺伝子の上流から 始まって、N, cIII, xis, int, sib
を含む領域が転写される。この様な RNA は sib
領域で2次構造をとるが、この2次構造が宿主の RNAaseⅢに認識され
xisの3'末ギリギリまでけずられ、intをコードする領域が無くなる。これにより、溶菌サイクルには不利な
int蛋白(ファージDNA の染色体DNAの組み込みに必要)は出来なくなる。これをretroregulationという。 |
|
 |
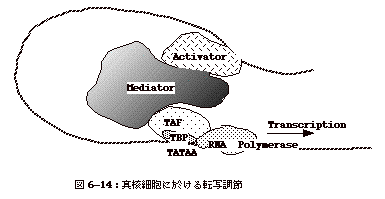
6-14:真核細胞に於ける転写調節
ここで、真核細胞の転写調節につき簡単に触れておく。
例えば、ヒトゲノムは100,000個の遺伝子を持つと推定されているが、このような多数の遺伝子を発生段階に応じて正確に発現させるには、微細な調節が必要であることが予想される。それには多数の調節因子が必要とされるであろう。実際、細菌とは異なり、多数の蛋白が転写に関っている。
大きく分けると3つのカテゴリーに分けられる。Activator、Mediator
complex、 RNA polymeraseを含む基本転写複合体(basal transcription complex)である(図6-14)。
Activatorは真核細胞には数百種あると云われている。遺伝子部位から可成り離れて存在するenhancer配列にActivatorは結合する。基本転写複合体はTBP(TATA
binding protein)、TAF (TBP-associated factor)、RNA polymeraseからなり、TBPはpromoter配列にあるTATAA配列を認識する役割を果たす。つまり、細菌のσ因子に似た働きをしている。
Activatorがpromoter部位に結合した基本転写複合体と相互作用し、転写が始まる訳であるが、直接相互作用する場合よりも、mediator或いはco-activatorが両者の相互作用を仲介することが多い。mediatorは20或いはそれ以上のペプチドからなる600-1,000kDa巨大な複合体である。この特徴は、(1)種々の異なったactivatorが一つのmediator複合体と相互作用し得ること、(2)異なったmediator間で同じsubunit蛋白をシェアしていること、である。同じsubunitを異なった複合体でシェア出来れば、限られた数(i番目のsubunitがNi種類あるとする)のsubunit蛋白でも、複数のsubunit(20とする)から出来ているので、何種類ものmediator複合体を作りえることとなる(N1
x N2 x N3 x ........N20)。こうして、各々の遺伝子に特異性のある調節が可能となる。又、同じmediator複合体をactivator間でシェア出来ることは、一つのactivatorにより異なった遺伝子発現を調節できる、つまり数多くの遺伝子を制御できると云うことである(Nature,
399,199-200 (1999))。 |
|
 |
| 当然の事であるが、これらの転写に関わる因子は細胞質で作られ、核に移行する。従って、核移行調節は転写調節の一段階となる。例えば、NFkBは細胞質でIFkBと結合しているが、特定のケモカインが細胞表面の受容対に結合すると、IFkBが燐酸化されubiquitinが付き分解する。するとIFkBが外れたNFkBは核へ移行し、特定の遺伝子の転写を誘導する。 Ubiquitination
ubiquitinが付くことをubiquitinationと呼ぶが、細胞内での蛋白の分解に必須な過程である。ubiquitinationには複数の酵素が関与し、ubiquitinの付いた蛋白はproteasomeと呼ばれる構造体の中で分解される。
細胞内の不要なシグナル(即ち、蛋白の酵素機能)を消失させる為にはその酵素を分解しなければならない。サイトカインで刺激された細胞を例にとると、その細胞でサイトカインのシグナルが伝達するには、直ちにIkBのシグナルを消さなければならない。細胞周期を考えると、細胞周期が上手く廻るには、特定の酵素蛋白がタイミングよくその時だけ機能する必要がある。その為には蛋白が合成されるだけでは不十分で、要らなくなったら直ちに無くなって貰わなければならない。この様に考えると、壊すと云う一見非生産的な過程が複数の酵素により調節を受けている事は理に叶った事である。
6-15: mRNAから蛋白への翻訳
mRNAの持つ情報を蛋白に読み取る場は、リボソームである。蛋白合成は、3つのステップからなる。開始(initiatioin)、伸長(elongation)、終止(termination)である。
最初に起こることは、特別な形のメチオニンの付いたfMet tRNA(N-formylethionyl
tRNA)とmRNAが30S ribosomeに付くことである。ここで、fMet tRNAはanticodonにより、mRNAの開始コドンAUGと結合している。そのリボソーム上の場所をP
site と云う。これに、50S subunitが付き伸長反応が始まる。アミノ酸の付いたtRNAがリボソームのA
(acceptor)siteにつきP (peptidyl)siteの伸長中のペプチドのC末アミノ酸と次々に重合する。これらの反応には複数の翻訳開始因子、翻訳伸長因子蛋白、エネルギー源としてのGTPが必要とされる。翻訳の停止は翻訳停止コドンによる。
リボソームは複数のリボソーム蛋白とリボソームRNAからなる(細菌の場合、蛋白は少なくとも50、RNAは3つ)。分子量も非常に大きい(細菌のリボソームの分子量は2.6x106ダルトン)。このリボソームを場として、伸長中のペプチド鎖にアミノ酸が順次転移し蛋白合成が起こる訳である。ここで起こる疑問は、この反応を行うのはどの分子かと云うことである。リボソームの蛋白か、RNAか、或いはそれ以外の酵素か、と云う問題である。最近、大腸菌のリボソームの構造解析から、この反応を触媒するのは蛋白ではなくリボソームの23SRNAが、そのリボザイム活性により、アミノ酸のペプチド鎖への転移を触媒するのではないか、と考えられ始めている。
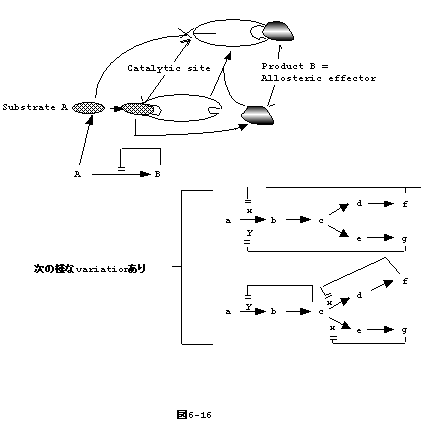
6-16:酵素活性調節(Regulation of Enzyme Activity)
上に述べたのは、mRNAの発現調節を介した酵素量の調節である。この他の調節としては、出来上がった酵素の活性を変えるものがある。上に述べた、IIIGlc
がリン酸化を受けIIIGlc-P となりadenylate cyclaseを活性化するのは、この一例である。
機構として二つある。
- Covalent modification:
例 phosphorylation, adenylation, acetylation, methylation
- Ligand binding, modulator binding
1.はリン酸化、アデニル化などによる酵素活性の変化である。
2.は、allosteric inhibitionとも呼ばれる。A の物質が酵素 E
と反応しB の物質が出来るとする。B が沢山溜ると E と反応し A
を認識させなくする。A と B
とは構造的に何等関係ないので、基質の競合ではない(図6-16) 。
考えてみるとA と B
とを遺伝的に結び付けるものはない。どうして、このような機構ができたのか、進化論的に謎である。
EmbdenーMeyerhof Pathway, TCA Cycleでもこのケースは多くみられる。
phosphoenolpyruvate は fructose-6-phosphate からfructoseー1,6-phosphate
への反応を抑制し、Acetyl-CoA は pyruvate から Acetyl-CoA への反応をallosteric
に抑制する。 |
|
 |
6-17:真核細胞に於ける蛋白の核膜輸送
細菌と異なり、真核細胞は核を持つ。即ち、核と細胞質の2つのコンパートメントを持つことになる。蛋白により、核、細胞質それぞれに局在する必要がある。これが上手く行かないと折角蛋白として発現しても機能しない。
例えば、リボソームRNA(rRNA)は転写されつつリボソーム蛋白と核内で会合し、リボゾームとなる。蛋白は総て細胞質で出来るので、リボソーム蛋白は先ず細胞質から核に移行する必要がある。一方出来上がったリボソームは細胞質で蛋白合成に使われる訳だから、核から細胞質に出て行かなければならない。このようにして、真核細胞では核と細胞質の間の蛋白の行き来が細胞機能上非常に重要となる。
次第に蛋白の核ー細胞質の行き来の機構が明らかになって来ており、図6-17はその模式図である。核膜には孔があり(nuclear
pore)、蛋白はここを通る。
| |
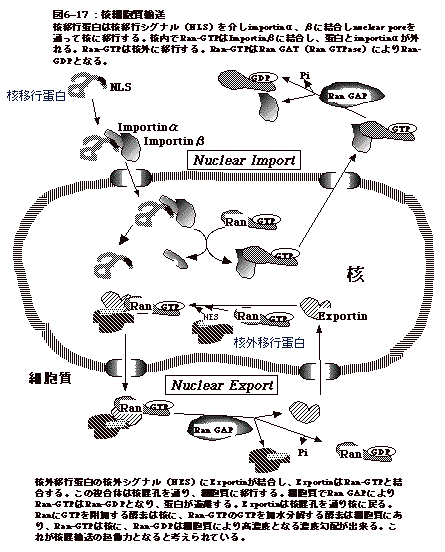 |
| 細胞質から核への移行にはimportin、核から細胞質への移行にはexportinが関係し、いずれの場合にもGTP結合型のRanが関与する。又、核移行蛋白にはnuclear
localization signal (NLS)、核から細胞質へ出る蛋白にはnuclear export signal
(NES)がある。 mRNAも少なくとも20種の蛋白の会合したhnRNP
(heterogenous nuclear RNA)の形で存在し、核から細胞質に移行する。恐らく、hnRNPの蛋白にNESに似たアミノ酸配列或いは構造があり、それが核外輸送の機構に認識され、蛋白との複合体として核外に出されるのであろう。 |
|
|
|
|
6-18:蛋白のスプライシング、intein
1990年頃、酵母の遺伝子発現を研究している途中で、コード領域が発現蛋白よりも遙かに大きいにも関わらず、RNA intronが見つからない現象に遭遇し、蛋白レベルで一部の切り出しがあるのではないか、と推定された。しかし、切り出し前に当たる高分子の蛋白が検出されないので信用されぬままであった。
1993年、Perler等が、除去されると推定される部分にあたる配列を別の蛋白に挿入し低温で精製し、その上で温度を上げてやると、この部分が切り出される事を報告した。以来沢山の報告が上がっていて、データベースInBase (www.neb.com/neb/inteins.html) が出来ている。
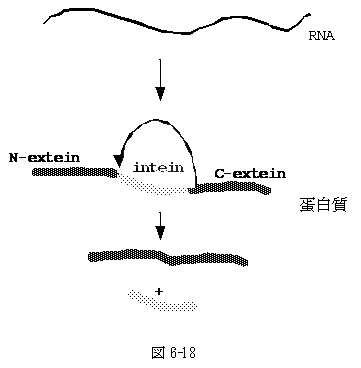
機構としては蛋白が畳み込まれ立体構造を取ると同時に自然にinteinのC末側配列(C-extein)がN末配列(N-extein)に取り付いて直ちにintein部分が外れると云うものである。温度やpHの変化でinteinの切り出しをコントロール出来るので、例えば、宿主に毒性のある蛋白を発現したい場合、その遺伝子の中にintein配列を挿入して発現させ、精製し、pHを変えてinteinを除去し目的の蛋白を精製するなど応用面も広い。 |
|
|
|
|